近日,中法植物表型国际联合实验室在遥感领域顶级期刊《Remote Sensing of Environment》在线发表了题为“Integrating prior information for improving 3D model-driven GAI estimation with application to wheat crops”的研究论文,提出“融合先验知识与三维数字孪生”的新策略,显著提升了跨生态点、多品种小麦全生育期监测的精度,为多站点联网表型监测提供了核心算法支撑。

开展涵盖多生态点、多品种的表型网络试验,并对作物生长发育过程进行高频、精准的遥感监测,是解析“基因-环境”互作机理、优化栽培管理和加速育种进程的关键。其中,绿色面积指数(GAI)作为表征作物光合作用器官表面积的核心指标,直接反映了作物的光合能力和生长状态。然而,由于传统表型算法对作物冠层结构差异、叶片生化特征变化以及土壤背景干扰等因素考虑不足,导致其在跨环境、多品种及全生育期的复杂应用场景下,监测误差显著增加,难以满足大范围、高精度表型网络监测的迫切需求。
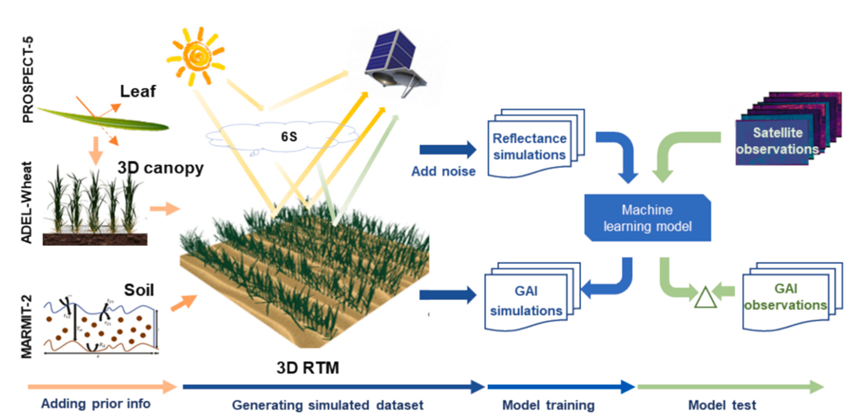

针对上述难题,本研究开发了一套增强模拟和反演框架:一是构建“数字孪生”底座——基于团队研发的数字化植物表型平台D3P,构建了小麦生长三维数字孪生体,实现了不同品种、土壤背景和光照变化下冠层反射率的精准模拟。二是融入广域“先验知识”——依靠国际合作网络的大量实测数据,将生物学规律提炼为“先验知识”植入数字孪生平台,极大提升了模型训练数据的代表性和真实度。三是分生育期“特异性建模”——针对小麦拔节前后的显著形态差异,分别构建了特异性反演模型,并进一步集成为全生育期组合模型,显著增强了模型的针对性与鲁棒性。
验证结果表明,相比传统方法,新模型在小麦GAI估算中取得了重要进展:预测准确性(R2)从0.82提升至0.93,均方根误差(RMSE)从0.73大幅降低至0.47。此外,全生育期组合模型在多生态点、多品种测试中表现出极高的稳定性。
本研究由南京农业大学前沿交叉研究院、教育部植物表型工程中心、中法植物表型国际联合实验室及作物遗传与种质创新利用国家重点实验室牵头开展。论文第一作者为董明霞博士,通讯作者为刘守阳教授。研究得到了南京农业大学丁艳锋教授,法国国家农业食品与环境研究院Frédéric Baret研究员与Marie Weiss研究员的重要指导。同时,法国作物学会Benoit de Solan研究员、日本东京大学郭威副教授与James Burridge副教授、加拿大自然资源部遥感中心Fernandes Richard研究员、南京农业大学姚霞教授、中国农业科学院农业资源与农业区划研究所李文娟研究员和农田灌溉研究所陈震副研究员等合作伙伴,提供了重要数据支撑。
原文链接:https://doi.org/10.1016/j.rse.2025.115161
数据和模型链接:https://github.com/PheniX-Lab/3DRTM-PlanetScope-GAI
阅读次数:12
【 转载本网文章请注明出处 】





